Eine neue Studie, die unter Leitung von Wissenschaftlern des Scripps Research Institute durchgeführt wurde, lässt den Schluss zu, dass der Replikationsvorgang der DNA – der genetischen Bauanleitung für alle lebenden Organismen, die sich aus den vier Basen C, G, T und A zusammensetzt – für unnatürliche „Buchstaben“ weitaus empfänglicher ist, als bisher gedacht wurde. Ein erweitertes „DNA-Alphabet“ könnte mehr Informationen erfassen als natürliche DNA, eine mögliche Kodierung für einen größeren Bereich von Molekülen sein und eine Vielfalt möglicher Anwendungsmöglichkeiten eröffnen, von präzisen molekularen Sonden und Nanomaschinen bis hin zu nützlichen neuen Lebensformen.
Die neue Studie, die in der aktuellen Ausgabe der Nature Chemical Biology vom 3. Juni 2012 erschienen ist, löst das Rätsel, wie ein zuvor entdecktes Paar künstlicher DNA-Basen den DNA-Replikationsprozess fast genauso effizient wie die vier natürlichen Basen durchwandern kann.
„Wir wissen, dass die effiziente Replikation unseres unnatürlichen Basenpaares kein Zufall ist und auch, dass der Replikationsprozess flexibler ist, als bisher angenommen“, sagte Floyd E. Romesberg, außerordentlicher Professor am Scripps Research Institute, leitender Entwickler der neuen DNA-Basen und einer der leitenden Autoren der neuen Studie. Das Romesberg-Labor arbeitete bei dieser neuen Untersuchung mit den Laboren der ebenfalls leitenden Autoren Andreas Marx von der Universität Konstanz und Tammy J. Dwyer von University of San Diego zusammen.
Erweiterung des DNA-Alphabets
Seit den späten 1990er Jahren suchten Romesberg und sein Labor nach einem Weg, das DNA-Alphabet zu erweitern. Im Jahr 2008 entwickelten sie die sich effizient replizierenden Basen NaM und 5SICS, die sich innerhalb der DNA-Helix wie ein komplementäres Basenpaar verhalten, so wie sich bei normaler DNA die Base Adenin (A) mit Thymin (T) verbindet und Cytosin (C) mit Guanin (G).
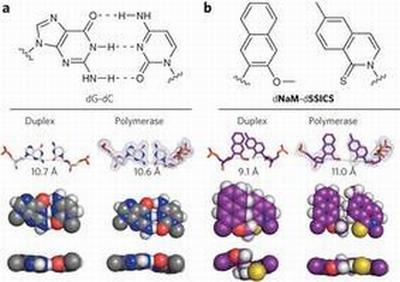
Im folgenden Jahr bewiesen Romesberg und seine Kollegen, dass NaM und 5SICS in der Petrischale effizient in RNA übertragen werden können. Aber der Erfolg dieser Basen bei der Imitation der Funktionen natürlicher Basen war etwas mysteriös. Sie wurden einfach nur dadurch gefunden, dass man tausende synthetische, Nukleotid-ähnliche Moleküle nach denen durchsucht hat, die am effizientesten repliziert wurden. Und es wurde sofort klar, dass ihrer chemischen Struktur die Fähigkeit fehlte, die Wasserstoff-Brückenbindungen zu bilden, welche die natürlichen Basenpaare in der DNA zusammenhalten. Man dachte, dass solche Bindungen absolut notwendig für eine erfolgreiche DNA-Replikation seien – ein Prozess, bei dem große Enzyme, die DNA-Polymerase, sich an einem einzelnen entwirrten DNA-Strang entlang bewegen und dabei den komplementären Strang quasi „anstricken“, immer eine komplementäre Base nach der anderen.
Eine frühere strukturelle Studie eines sehr ähnlichen Basenpaares in der DNA-Doppelhelix kam noch zu Romesbergs Bedenken hinzu. Die Daten deuteten stark darauf hin, dass NaM und 5SICS sich nicht auch nur annähernd in die nahtlose Geometrie natürlicher Basenpaare einfügen, welche nach den beiden Entdeckern der DNA-Doppelhelix Watson-Crick-Geometrie genannt wird. Stattdessen verbinden sie sich auf eine lockerere, überlappende „interpolierende“ Weise. „Ihre Paarung ergibt ‚Unpaare‘, wie etwa zwei identische Basen zusammen, die normalerweise von der DNA-Polymerase nicht als gültiges Basenpaar erkannt werden würden“, so Denis Malyshev, Doktorand in Romesbergs Labor und zusammen mit Karin Betz aus dem Labor von Marx leitender Autor.
Dennoch war das NaM-5SICS-Basenpaar Test für Test ausreichend replizierbar. „Wir fragten uns, ob wir die DNA-Polymerase irgendwie austricksten, so dass sie erkannt wurden“, sagte Romesberg. „Ich wollte die Entwicklung von Anwendungen nicht weiter fortsetzen, bevor wir nicht ein klareres Bild davon hatten, was bei der Replikation vor sich ging.“
Rand an Rand
Um dieses klarere Bild zu erhalten, wandten sich Romesberg und sein Labor an die Labors von Dwyer und Marx, die Expertenwissen beim Herausfinden der atomaren Strukturen von DNA in Verbindung mit DNA-Polymerase hatten. Ihre strukturellen Daten zeigten nur, dass das NaM-5SICS-Paar innerhalb der DNA-Doppelhelix seine abnormale, interpolierende Struktur beibehielt – aber dennoch die normale, fugenlose Watson-Crick-Position einnahm, wenn es im kritischen Moment der DNA-Replikation von der Polymerase „ergriffen“ wurde.
„Die DNA-Polymerase veranlasste offenbar das unnatürliche Basenpaar dazu, eine Struktur einzunehmen, die virtuell nicht von der Struktur natürlicher Basenpaare zu unterscheiden ist“, sagte Malyshev.
NaM und 5SICS, denen die Wasserstoff-Brückenbindungen fehlen, werden innerhalb der DNA-Doppelhelix von „hydrophoben“ Kräften zusammenhalten. Solche Kräfte verursachen, dass bestimmte molekulare Strukturen (wie zum Beispiel Öl) von Wassermolekülen abgestoßen werden und sich deshalb in einem Wassermedium miteinander verbinden. „Es ist durchaus möglich, dass diese hydrophoben Kräfte Charakteristika besitzen, die die Flexibilität und dadurch auch die Replizierbarkeit des NaM-5SICS-Basenpaares ermöglichen“, sagte Romesberg. „Sicherlich wären sie nicht fähig, während der DNA-Replikation in die richtige Form zu springen, wenn ihre abweichende Struktur in der Doppelhelix von stärkeren kovalenten Bindungen zusammengehalten werden würde.“
Eine willkürliche Wahl?
Die Ergebnisse legen nahe, dass NaM-5SICS und möglicherweise andere, hydrophob verbundene Basenpaare eines Tages das Alphabet der DNA erweitern könnten. Sie weisen ebenfalls darauf hin, dass die Wahl der Evolution für das bestehende, vier Buchstaben umfassende DNA-Alphabet – auf unserem Planeten – vielleicht ein bisschen zufällig war. „Es sieht so aus, als könnte das Leben auch auf vielen anderen genetischen Systemen basiert haben“, sagte Romesberg.
Seine Laborkollegen und er versuchen jetzt, die grundlegende Funktionalität von NaM und 5SICS zu optimieren und zu zeigen, dass diese neuen Basen auch zusammen mit den natürlichen Basen in der DNA einer lebenden Zelle arbeiten können.
„Wenn wir das neue Basenpaar dazu bringen können, sich mit hoher Effizienz und Genauigkeit in vivo zu replizieren, haben wir einen halbsynthethischen Organismus“, so Romesberg. „Die Dinge, die man damit anstellen könnte, sind ziemlich umwerfend.“
Weitere Beitragende zur Abhandlung „KlenTaq polymerase replicates unnatural base pairs by inducing a Watson-Crick geometry“ sind Thomas Lavergne vom Romesberg Labor, Wolfram Welte und Kay Diederichs vom Marx Labor und Phillip Ordoukhanian vom Center for Protein and Nucleic Acid Research am The Scripps Research Institute.
Die Studie wurde in Teilen durch ein Stipendium der National Institutes of Health finanziert.
Quelle: http://www.scripps.edu/news/press/2012/20120603romesberg.html
(SOM)
Antworten